基于人工智能的通用蛋白质工程方法成功开发
军工资源网 2025年07月08日蛋白质工程基于蛋白质的灵活性,通过人工手段改变氨基酸序列,实现对蛋白质结构和功能的修饰和改造。与基因组工程相比,蛋白质工程可直接对蛋白质分子进行操纵,借助突变的迭代积累,快速完成蛋白功能优化和创新。
蛋白质工程改造策略包括结构引导的蛋白质理性设计和定向进化,但这些方法往往依赖经验,存在实验周期长、成本高等问题,限制了规模化应用。理想的蛋白质工程策略应能够以最小投入实现最优工程性能。当前,通过训练特定蛋白专有的人工智能模型实现突变模拟和功能改造是蛋白质工程新方向。但是,这些模型在拓展应用到多种蛋白时存在困难,面临通用性欠佳问题;同时,模型训练和下游验证需要大量的计算和实验成本,限制了应用。因此,亟需开发高效、普适且无需复杂模型训练的蛋白质工程计算模拟策略,以最大限度地减少计算负荷、实现最大化性能。
近日,中国科学院遗传与发育生物学研究所高彩霞团队基于整合的结构与进化约束的通用逆折叠模型,开发出新型人工智能蛋白质工程计算模拟方法AiCE(AI-informed Constraints for protein Engineering)。这一方法无需训练专属人工智能模型,即可实现蛋白质高效进化模拟和功能设计。该团队利用AiCE对多种基因编辑工具进行进化优化,实现了效率和精度的快速提升。
蛋白质逆折叠是利用AI模型,通过给定三维结构预测可兼容序列的过程。通用的蛋白质逆折叠模型通过天然蛋白质结构和序列的训练,可以隐式学习蛋白质骨架的几何和物理特性,捕捉由进化动力学塑造的蛋白质序列的复杂分布模式。
该团队基于现有通用逆折叠模型开发出AiCEsingle模块。具体来说,该团队基于给定的蛋白质三维结构,对逆折叠模型输出的氨基酸序列开展采样,来提名高频出现的氨基酸类型,进一步通过结构约束对氨基酸频率开展差异筛选,得到最终预测的单个氨基酸替换类型。团队利用60个深度突变扫描数据,测试AiCEsingle性能,发现其实现了16%的预测准确率;通过消融实验和逻辑回归分析,证明结构限制在方法中的必要性,相比于无限制方案性能提升了37%。进一步,平行比较分析显示,AiCEsingle相比于其他常见AI模型实现了36%至90%以上的性能提升。从蛋白类型来看,AiCEsingle实现了复杂蛋白和蛋白质-核酸复合物如CRISPR蛋白、SARS-CoV-2病毒蛋白等的有效进化,具有广泛的通用性。为克服突变组合广泛存在的负向上位效应,团队假设存在进化耦合的氨基酸位置可能存在功能协同,构建了通过预测进化耦合性来预测突变组合位置的AiCEmulti模块。6个突变文库的分析结果表明,AiCEmulti与蛋白质大模型SaProt预测能力相当,但计算成本极低。
该团队建立的包含两类模块的AiCE方法,可实现单突和组合突变的快速有效预测。这一方法利用现有的通用逆折叠模型而无需重新/迁移训练专有蛋白模型,降低了计算成本,只需1.15个CPU时即可识别SpCas9蛋白的单突和双突变体。
进一步,利用这一方法,该团队在湿实验层面实现了包括脱氨酶、核定位序列、核酸酶和逆转录酶等8种结构和功能多样蛋白质的AiCE功能验证,证明了其简单、高效和通用性。借助优化的脱氨酶,团队开发了可用于精准医疗和分子育种的新型碱基编辑器,包括编辑窗口缩小近一半的新型胞嘧啶碱基编辑器enABE8e、保真度提升1.3倍的新型腺嘌呤碱基编辑器enSdd6-CBE以及活性提升13倍的新型线粒体碱基编辑器enDdd1-DdCBE。
研究发现,与传统蛋白质工程方案相比,AiCE在效率、可扩展性和通用性方面均具有优势。
7月7日,相关研究成果以Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints为题,发表在《细胞》(Cell)上。研究工作得到农业农村部相关项目、国家自然科学基金、国家重点研发计划等的支持。
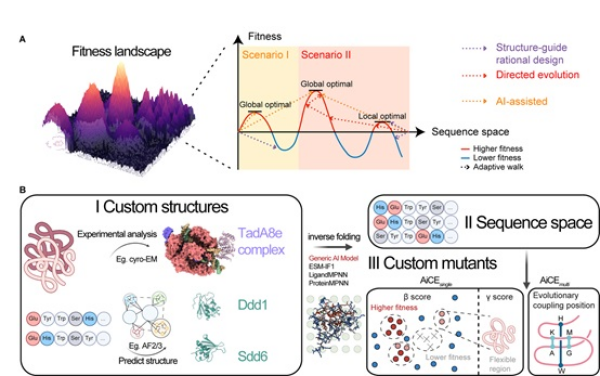
常见蛋白质工程方法的示意图和AiCE方法概述
